1 实验流程
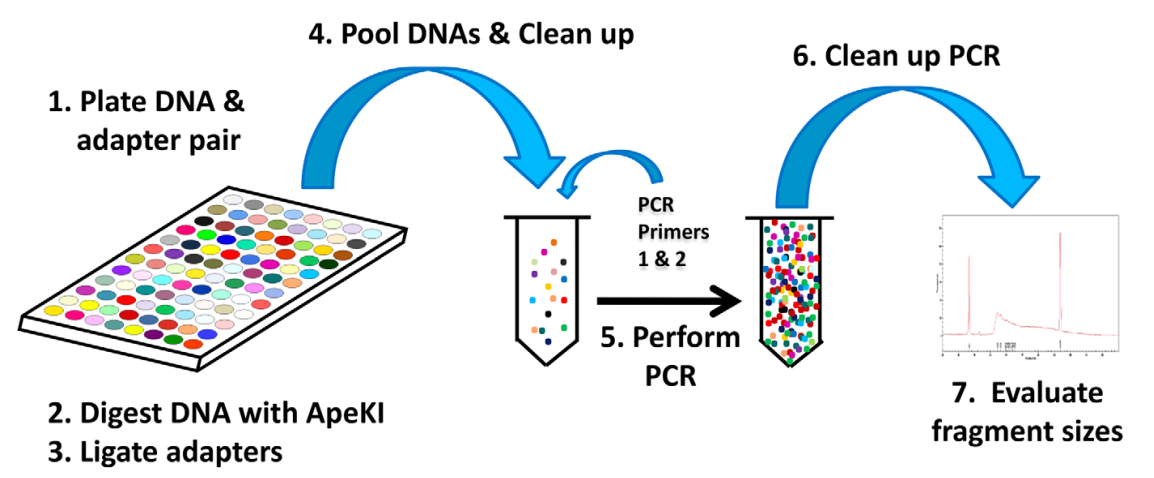
图1 实验流程
2 生物信息分析
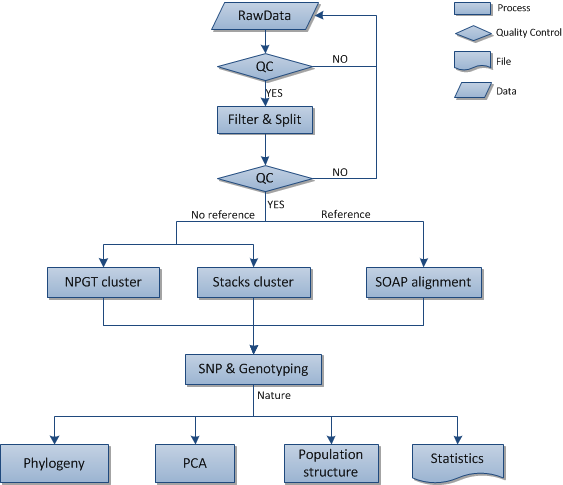
图2 标准信息分析流程
2.1 数据统计
此项目根据合同要求,共产出52.61 Gb数据,其中部分样品的数据量统计如下(各列含义请见Data.readme.txt),这里仅取部分文件示意:
| Sample name | Read number (M) | Base number (Mb) | GC (%) | Q20 (%) | Q30 (%) |
|---|---|---|---|---|---|
| 1HL_10A | 6.37 | 615.01 | 42.61 | 95.68 | 90.80 |
| 1HL_11A | 7.14 | 688.72 | 42.69 | 95.72 | 90.91 |
| 1HL_12A | 5.05 | 487.29 | 42.57 | 95.61 | 90.70 |
| 1HL_13A | 5.35 | 516.55 | 42.84 | 95.63 | 90.72 |
| 1HL_14A | 4.03 | 387.00 | 42.69 | 95.59 | 90.64 |
| 1HL_15A | 4.39 | 421.69 | 42.50 | 95.59 | 90.62 |
| 1HL_16A | 4.67 | 447.97 | 42.62 | 95.63 | 90.74 |
| 1HL_17A | 7.31 | 701.52 | 42.51 | 95.61 | 90.70 |
| 1HL_19A | 7.24 | 694.65 | 42.76 | 95.64 | 90.73 |
| 1HL_1A | 4.52 | 436.26 | 42.36 | 95.61 | 90.66 |
| ... | ... | ... | ... | ... | ... |
* 所有样品数据统计列表Data.stat.xls
2.2 SNP结果
下列表格为SNP统计信息(各列含义详见SNP.readme.txt),这里仅取部分文件示意:
| Sample name | Total | Homo | Hete | Homo rate (%) | Hete rate (%) |
|---|---|---|---|---|---|
| 1HL_10A | 8058 | 6755 | 1303 | 83.83 | 16.17 |
| 1HL_11A | 8392 | 7033 | 1359 | 83.81 | 16.19 |
| 1HL_12A | 7422 | 6405 | 1017 | 86.30 | 13.70 |
| 1HL_13A | 7580 | 6499 | 1081 | 85.74 | 14.26 |
| 1HL_14A | 6374 | 5552 | 822 | 87.10 | 12.90 |
| 1HL_15A | 6683 | 5706 | 977 | 85.38 | 14.62 |
| 1HL_16A | 7041 | 6043 | 998 | 85.83 | 14.17 |
| 1HL_17A | 8419 | 7050 | 1369 | 83.74 | 16.26 |
| 1HL_19A | 8543 | 7141 | 1402 | 83.59 | 16.41 |
| 1HL_1A | 6919 | 5949 | 970 | 85.98 | 14.02 |
| ... | ... | ... | ... | ... | ... |
* 所有样品的SNP统计表见SNP.stat.xls
2.3 基因分型
下列表格为基因分型统计信息(各列含义详见genotype.noref.readme.txt),这里仅取部分文件示意:
| ID | Consensus_Seq | pos | 1SN_9A | 1SN_8A | 1SN_7A |
|---|---|---|---|---|---|
| record_385 | CAGCCCTTATTAGGCCACCTGAGTRAACTGATGTGACCTATATTGTAATTAGTTTTCATCCATCATTGTTAAGTCATATGTA | 25 | A | A | A |
| record_795 | CAGCTTCCGATGCGTCGGAGAAGATCAGCACTTYTGGATCTGATACTAAGCTCAGTGATTGTGGAACAAACATCCGTGGAAT | 34 | Y | Y | Y |
| record_1907 | CAGCTGAAGCATCCAGCAAAGCACGAACAAAGCAGAGAGACACCTACAACACCAAAGTGAGARGAGGAACGGTTAAGGCTGG | 63 | G | G | - |
| record_2412 | CAGCCCTTCACCGGTAATGGTGACGTCTCAATATGAATGAAATATTCTCGACGGGWCGAAAAACAACAAATAATCAATCAAT | 56 | - | - | - |
| record_3293 | CAGCACCTCAGGGCTATCCGTTTCTAACACGGGTACGGGCCMGCGGGGGTTCACGGGCAGTGACCTCCCTTGCACGTTACAA | 42 | - | C | - |
| record_4526 | CTGCACTTGACCACAATAAATGTGATCTTCCTTTAGATTTAATATATATCTGTCTGCCTTCATRAACGTTGATTTTTAAAAT | 64 | R | G | R |
| record_4591 | CAGCTGTATCATCTGACCCCAGAAGAAGCCGGYTTGATAGGAAGCTCTCTTGATGTGTGCTTCTCGAGTTGGAGGAATGGCC | 33 | C | C | C |
| record_5127 | CTGCGTTGTCATGCAAGCATTCTTGACTCCCATGCTCACGAGCCATAGTTACAATGCCGGAATCGCRCATTGGTTTATTCGC | 67 | R | G | R |
| record_5132 | CAGCTTAAGATTTTGTGCATTTTTCAGTTGTGYTCTCTCTACTTTCGATTCCTTGATCCGCCCTTGAAGGAGACCTTCATGT | 33 | C | C | - |
| record_5456 | CAGCTTGTTTACGAGGCCCTGCYCGCACTTTCTACATAAGTCTCCCCGCAGAATTCAGACAGCCATATAACTTGTTAATTAC | 23 | Y | Y | Y |
| ... | ... | ... | ... | ... | ... |
* 所有样品的基因分型结果Genotype.xls
2.4 系统发育分析
根据得到的SNP信息,我们可以推断出种群间的进化关系,并用分支图表示,结果请见下图:

图3 系统发育树.
2.5 主成分分析
主成分分析(Principal Component Analysis, PCA)是指通过线性变换找出数据集中对方差贡献最大的主要成分,以降低数据复杂度的一种分析。基于个体间的SNP差异程度,利用PCA按照不同性状特征可以将样本按主成分分类成不同的亚群,分析结果请见下图:
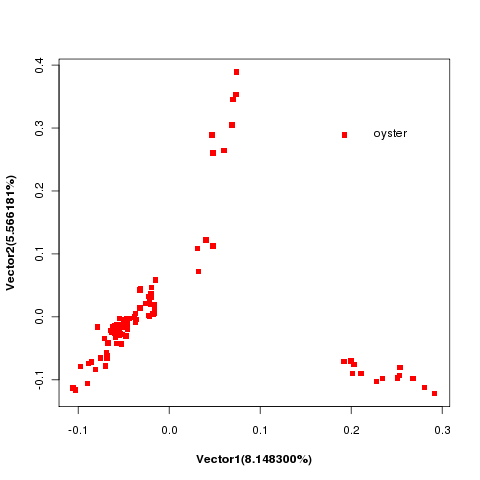
图4 PCA分析.
注:不同颜色的点代表不同亚群的样本。